基於結構預測序列,James Collins開發RNA適配體從頭設計模型
RNA 能夠執行多種生物功能。
基於計算機技術和人工智能算法,包括 RhoFold、trRosettaRNA、DeepFoldRNA 和 AlphaFold3 等在內的幾種基於深度學習的方法已經能夠完全自動化地從序列預測 RNA 3D 結構。
近日,麻省理工學院 James Collins 團隊開發了一個基於結構預測序列的深度學習平台 RhoDesign,用於 RNA 適配體的從頭設計。這種方法可以設計出結構相似但序列不同的 RNA 適配體,能夠在小分子存在下產生螢光。
相關文章以題為「Deep generative design of RNA aptamers using structural predictions」發表在 Nature Computational Science 期刊。James Collins 實驗室的博士後 Felix Wong、香港中文大學計算機科學及工程系 Dongchen He 是本研究共同一作。Felix Wong 也是 Integrated Biosciences 的聯合創始人,這是一家致力於研發與年齡相關疾病藥物的新興生物技術初創公司。
RNA 適配體因其結構的多樣性而具有靶分子廣、親和力高、特異性強等特點;同時,相比傳統抗體,RNA 適配體分子量小、易改造修飾、製備方便且無免疫原性。因此,RNA 適配體在基礎研究、臨床診斷、藥物研製等方面展現了廣闊的應用前景。
RhoDesign 建立在 RhoFold 基礎之上,後者在與其他結構預測模型對比時展現出有前景的性能。RhoFold 是由智峪生科團隊主導,並聯合港中文和複旦大學團隊共同完成的全球第一款全自動的端到端 RNA 3D 結構預測深度學習模型。
首先,研究人員整理了 369,499 個由 RhoFold 預測的 3D RNA 結構,以及從蛋白質數據銀行(PDB)中獲取的 3,435 個 3D RNA 結構,並使用這些結構及其對應的序列來訓練深度學習模型;另外,研究人員額外提供了與 3D 結構對應的預測或真實二級結構以提高準確性。
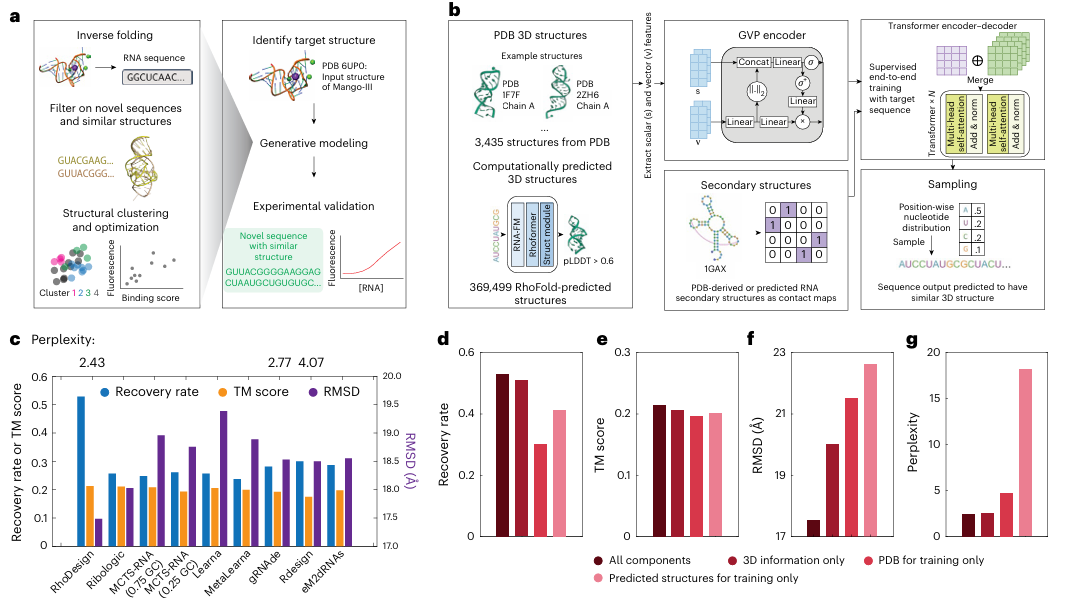 圖 | 一種基於 3D 結構的 RNA 生成設計深度學習方法
圖 | 一種基於 3D 結構的 RNA 生成設計深度學習方法在模型訓練後,研究人員使用恢復率、模板建模(TM)分數、均方根偏差(RMSD)和困惑度來對訓練模型進行基準測試。
結果發現,與包括 LEARNA、Meta-LEARNA、RiboLogic、gRNAde、RDesign 和 eM2dRNAs 等在內的模型相比,RhoDesign 的表現更優。
基準測試後,應用 RhoDesign 來從結構生成 RNA 序列。
與小分子 TO1-biotin 結合的 RNA 適配體 Mango 已經被廣泛表徵。在這裏,作者考慮了 PDB 6UP0 中的 Mango-III (A10U)適配體。PDB 6UP0 是 PDB 中的一個條目,它包含了 Mango-III 螢光 RNA 適配體與 YO3-biotin 結合物的三維結構。
首先,他們重新訓練 RhoDesign 以排除與 6UP0 中的 Mango-III 適配體序列相似度大於 0.5 的結構,以測試模型的泛化能力;然後,將訓練好的模型應用於使用 6UP0 的結構作為輸入生成 60 個候選序列,這些序列一致地被 RhoFold 很好地預測。
從生成的序列中,篩選出了 18 個符合條件的適配體進行評估。作為比較,研究人員還分別使用 RaptGen 和其他 7 個從結構到序列生成模型合成了 22 個和 90 個適配體。
在測試合成的適配體在 TO1-biotin(10 微米)存在下的螢光時,發現 18 個 RhoDesign 生成的適配體中有 4 個表現出活性。22 個 RaptGen 生成的適配體中有 20 個也表現出活性,但這些序列與任何 Mango 適配體的最大序列相似度都大於 0.7。
相比之下,4 個活躍的 RhoDesign 生成的適配體與任何 Mango 適配體的最大序列相似度為0.59;其中,適配體 1 更引人注目,它顯示出比Mango-I 更高的螢光。從其他 7 個結構到序列生成模型中合成的 70 個適配體中只有 4 個表現出活性;其中,1 個是 gRNAde 生成的,3 個是 MCTS-RNA 生成的。後者的工作命中率為 30%,略高於 RhoDesign 的工作命中率(22%)。
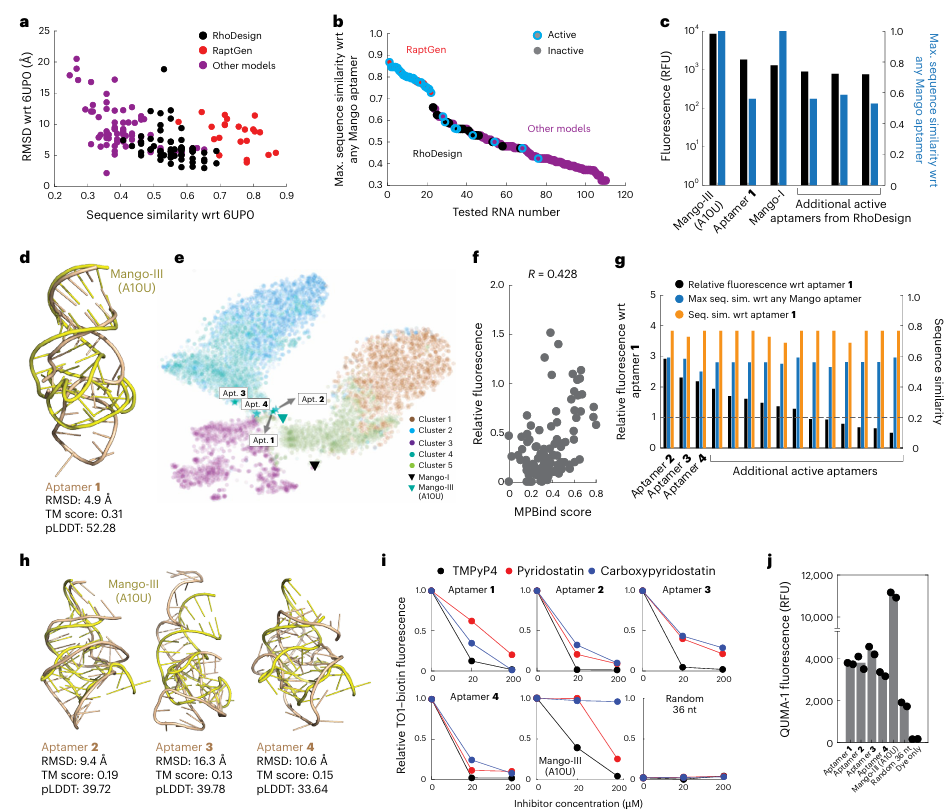 圖 | 生成的 RNA 適配體的螢光實驗驗證、優化和機制研究
圖 | 生成的 RNA 適配體的螢光實驗驗證、優化和機制研究然而,其他方法生成的 4 個活躍適配體都沒有顯示出比適配體 1 更強的螢光,並且 RhoDesign 在基準測試中顯示出大約是 MCTS-RNA 的兩倍的恢復率。儘管適配體 1 預測與 Mango-III 結構相似,但它不包含已知的 Mango 適配體螢光活性的基礎保守序列基序,這表明它可能是一個前所未有的具有 Mango 樣活性的適配體。
為了進一步優化適配體 1,將適配體 1 的 RhoFold 預測結構作為輸入提供給 RhoDesign,生成了一組適配體1衍生物。並選擇了活性更高的前三名適配體 2-4 進一步研究。
結果發現,適配體 2-4 的螢光強度大於適配體 1。它們的預測 3D 結構,儘管與 Mango-III 的結構相似度比適配體 1 的低,但差異很小,表明它們可能部分通過與 Mango 適配體相似的機制發光。
總而言之,這項研究提供了一個基於計算機的 RNA 序列從頭設計平台。與其他平台相比,其可以促進活性 RNA 適配體的結構引導設計,預計將這些方法與 RhoDesign 集成可以進一步提高 RNA 設計的準確性。鑒於當前基準測試的局限性,RhoDesign 也將從使用更可靠的 RNA 3D 結構預測方法進行進一步驗證以及未來與多樣化 RNA 的額外實驗測試中受益。
參考鏈接:
1.Wong, F., He, D., Krishnan, A. et al. Deep generative design of RNA aptamers using structural predictions. Nat Comput Sci (2024). https://doi.org/10.1038/s43588-024-00720-6