蛋白質設計再突破!麻省理工團隊利用人工智能與梯度優化開發新策略
蛋白質在我們的身體中扮演著構建結構、催化化學反應、運輸物質以及識別和抵禦病原體等關鍵角色。它們在生長髮育、新陳代謝和免疫防禦中發揮著不可替代的作用。如今,科學家們不僅滿足於自然界中的蛋白質,還試圖創造全新的蛋白質以滿足特定需求,如設計能夠精準識別並中和特定病毒的蛋白質用於疾病治療,或打造高效的生物催化劑以加速工業生產中的化學反應。
機器學習通過學習已知蛋白質功能數據,能夠建立蛋白質結構與功能之間的複雜關係模型,為蛋白質的精確設計帶來無限可能。
2024 年,蛋白質設計領域迎來了里程碑——蛋白質從頭設計的先驅大衛·貝克,以及軟件 Alphafold2 的開發者 Demis Hassabis 和 John M. Jumper 被授予盧保化學獎。Alphafold2 的出現極大地提高了蛋白質結構預測的精度,為蛋白質設計帶來了革命性的變化。
在此基礎上,由慕尼黑工業大學的 Hendrik Dietz 教授和麻省理工學院的 Sergey Ovchinnikov 教授領導的國際研究團隊,進一步拓展了蛋白質設計的邊界。他們將 Alphafold2 的精確結構預測能力與梯度下降優化方法相結合,開發出了一種創新的蛋白質設計方法。該研究成果發表在頂級學術期刊 Science 上。
梯度下降是模型優化的常用方法,可以逐步識別與目標函數的偏差,並調整參數,直至達到最優結果。在蛋白質設計中應用梯度下降,可以將 AlphaFold2 預測的新蛋白質結構與所需的蛋白質結構進行比較,並使科學家得以進一步優化他們新設計的氨基酸鏈和由此產生的結構,後者在很大程度上決定了蛋白質的穩定性和功能。
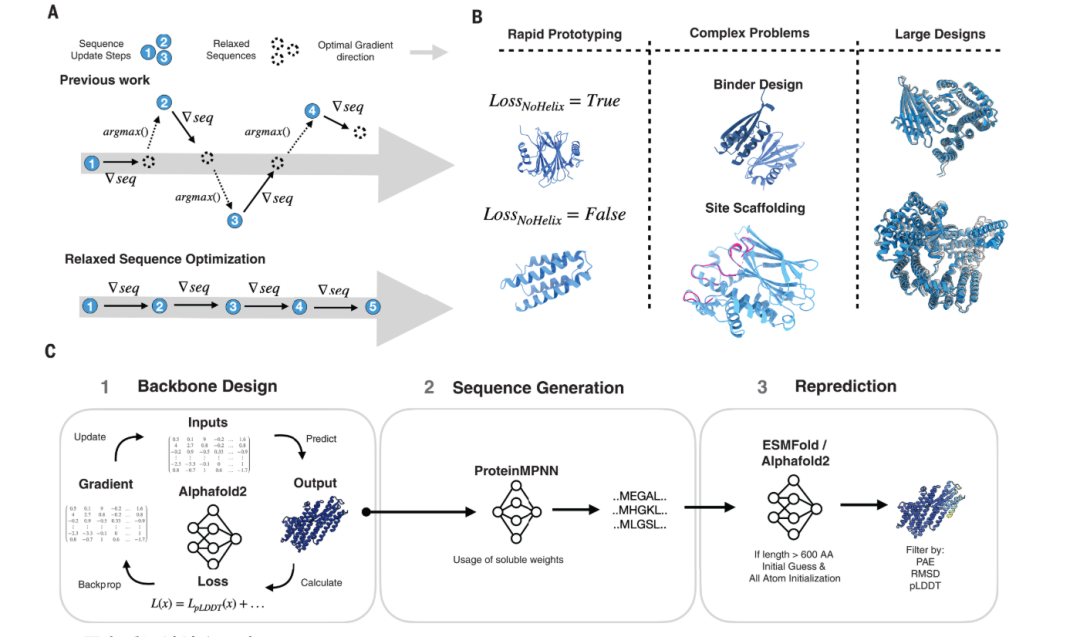 圖|人工蛋白質設計流程示意圖
圖|人工蛋白質設計流程示意圖該方法的獨特之處在於,它打破了傳統設計方法的局限。以往在設計蛋白質時,通常會假設氨基酸鏈的每個位置只能由 20 種天然氨基酸中的一種佔據。而新方法則採用了一種更為靈活的策略,在設計初期,它允許所有可能的氨基酸在每個位置上以一種虛擬疊加的狀態存在,為蛋白質的優化提供了豐富的選擇。通過多次迭代優化,這些虛擬疊加的氨基酸逐漸「凝聚」成一個最接近目標結構的穩定結構。此時,研究人員就可以根據這個優化後的結構,確定出能夠在實驗室中實際合成蛋白質的具體氨基酸序列,從而將虛擬的設計轉化為現實的蛋白質分子。
目前,研究團隊利用新方法在計算機上虛擬設計了 100 多種蛋白質,這些蛋白質各具獨特的功能和特性,如模擬酶的催化活性、模擬抗體與抗原的結合特異性等。隨後,他們在實驗室中成功合成了這些蛋白質,並對其進行了一系列嚴格的實驗測試。
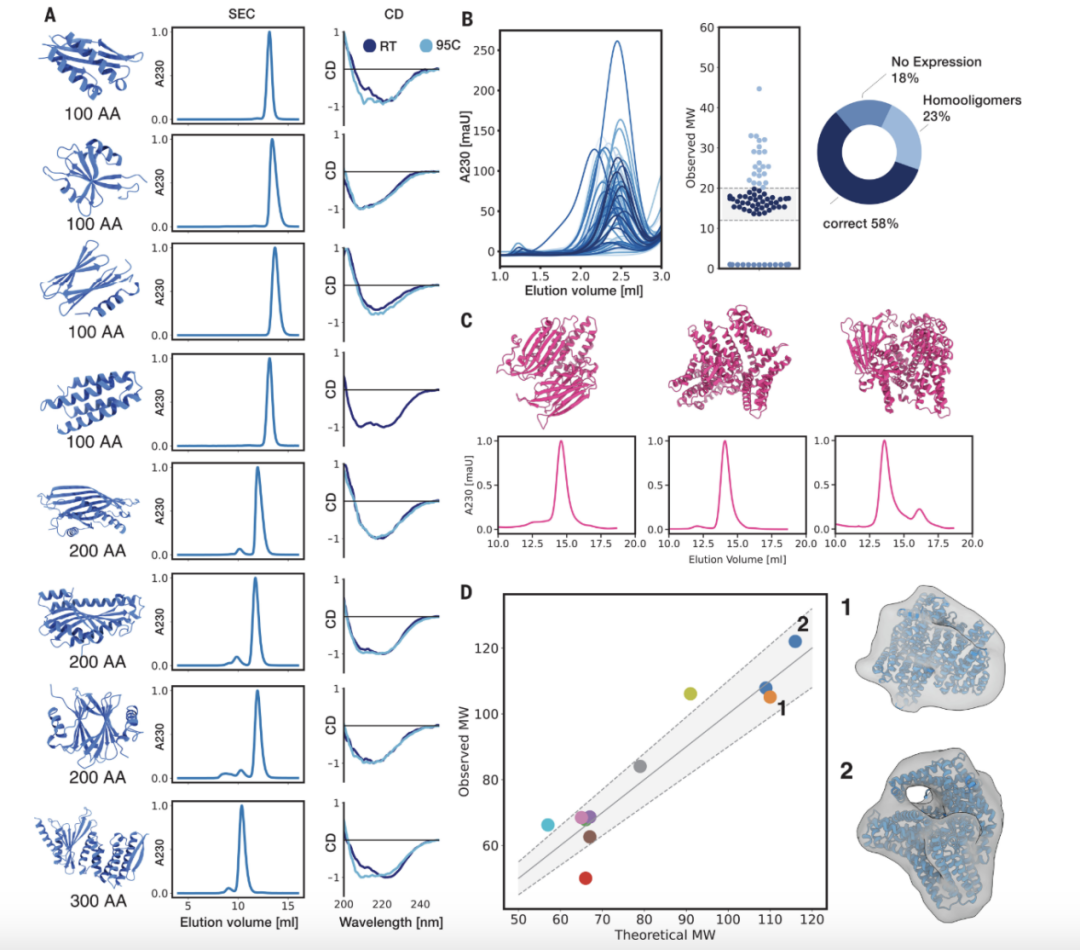 圖|實際生產蛋白質與計算機預期結構表現出高度相符
圖|實際生產蛋白質與計算機預期結構表現出高度相符實驗結果令人振奮——實際生產出的蛋白質結構與計算機預測的結構高度吻合。研究人員對其中 9 種單體蛋白質進行親和色譜純化,通過天然尺寸排阻色譜(SEC)分析,8 種表達良好且在預期分子量分數處有單一主峰;另外 76 種蛋白質採用高通量表達和純化策略,58% 的蛋白質分子量與 SEC 校準預期相符。
例如,通過X射線衍射和雪藏電鏡等技術確定的蛋白質結構,其與設計模型的低 RMSD 值(如 200 個氨基酸的蛋白質 Ca-RMSD 為 0.90 Å、400 個氨基酸的為 1.28 Å、600 個氨基酸的為 0.92 Å 等)表明了其結構的準確性,並有著實現預期功能的潛力。
不僅如此,研究人員還利用新方法設計出由多達 1000 個氨基酸組成的大型蛋白質,這令人工設計的蛋白質在大小上能夠更接近天然存在的抗體,意味著我們可以像在抗體中整合多種功能一樣,將不同的功能模塊巧妙地組合到這些人工蛋白質中。例如,我們可以設計出一種既能精準識別癌細胞,又能攜帶藥物分子並在特定部位釋放的蛋白質,為癌症治療帶來新的希望。
總而言之,該研究成果為蛋白質設計領域帶來了新的曙光,它將加速我們對蛋白質結構與功能關係的理解,推動蛋白質設計在生物醫學、生物工程和材料科學等眾多領域的廣泛應用。或許在不久的將來,我們將見證更多基於這些人工設計蛋白質的創新療法和技術的誕生。
參考文獻:
1. Frank C, Khoshouei A, Fuβ L, Schiwietz D, Putz D, Weber L, Zhao Z, Hattori M, Feng S, de Stigter Y, Ovchinnikov S, Dietz H. Scalable protein design using optimization in a relaxed sequence space. Science. 2024 Oct 25;386(6720):439-445. doi: 10.1126/science.adq1741. Epub 2024 Oct 24. PMID: 39446959.