從0編寫基因組!史上最大生物學模型Evo-2全面開源:矽基生命能創造細胞?

新智元報導
編輯:LRS
【新智元導讀】史上最大的基因組AI模型Evo 2使用超過12.8萬個基因組數據訓練,包含9.3萬億個核苷酸,能預測突變效應、設計 DNA 序列,並通過可視化工具展示學習到的生物特徵,為生成生物學和疾病研究提供新思路。
生命的一切表現,基本都可以從DNA編碼中找到答案。
基因組(Genome)包含了生物體所有基因以及非編碼的DNA序列,承載了生物體發育、生長、繁殖和適應環境所需的全部遺傳信息,近年來基因組相關的測序、合成和編輯工具已經徹底改變了生物學研究。
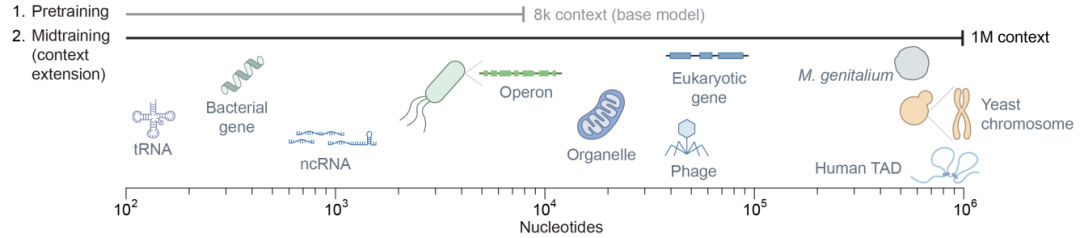
然而,基因組的複雜性是巨大的,即使是最簡單的微生物,也包含數百萬個DNA堿基對,要智能地構建新的生物系統,研究人員還需要深入理解基因組編碼的複雜信息。
2024年11月,研究人員在Science上發表了一項研究Evo 1,基於單細胞(270萬個原核生物和噬菌體)基因組進行訓練,具有70億個參數,在單核苷酸(構成DNA或RNA的基本單元)設置下實現了13萬堿基的上下文長度。

Evo 1論文鏈接:https://www.science.org/doi/10.1126/science.ado9336
Evo 1在DNA、RNA 和蛋白質模式上展示了更好的零樣本功能預測能力,並通過實驗驗證了Evo 1生成的CRISPR-Cas分子復合物以及IS200和IS605轉座系統的功能活性,證明了使用語言模型進行蛋白質-RNA和蛋白質-DNA代碼設計的前景。
最近,Arc Institute宣佈與英偉達(NVIDIA)合作,開發了迄今為止最大的生物領域AI模型Evo 2,在超過12.8萬個全基因組以及宏□因組數據的基礎上,訓練數據超過9.3萬億個核苷酸。
除了細菌、古菌和噬菌體基因組外,Evo 2的訓練數據還包括人類、植物以及其他真核生物域的單細胞和多細胞物種的信息。

論文鏈接:https://www.biorxiv.org/content/10.1101/2025.02.18.638918v1
Evo Designer鏈接:https://arcinstitute.org/tools/evo/evo-mech-interp
隨文章一起發佈的,還有一個Evo Designer的用戶友好界面,作為機制可解釋性可視化器,揭示了模型在基因組序列中學習的關鍵生物特徵和模式,展示了Evo 2在生成DNA序列時的思考過程,其中許多功能與基因組概念類別高度相關,用戶可以在參考基因組和Evo 2中的相應特徵激活中看到基因組概念。
研究人員完全開源了Evo 2的訓練數據、訓練和推理代碼以及模型權重,並集成到了NVIDIA BioNeMo框架中。
考慮到潛在的倫理和安全風險,研究人員將感染人類和其他複雜生物體的病原體排除在Evo 2的基礎數據集之外,並確保模型不會對關於這些病原體的查詢返回有價值的答案。
Arc研究所聯合創始人、Arc核心研究員、加州大學伯克利分校生物工程助理教授Patrick Hsu認為,Evo 1和Evo 2的成功是「生成生物學」領域的關鍵時刻,讓機器也能夠用核苷酸的語言進行閱讀、寫作和思考,Evo 2對生命之樹具有通才式的理解,對於很多任務來說都有價值,比如預測致病突變、設計人工生命的潛在編碼。
Evo 2及其後續版本是基因組和表觀基因組設計領域邁向生成式生物學的第一步,結合現有的、在大規模可編程DNA操作方面的最新實驗進展,Evo 2有望實現多樣化合成生命的直接編程。

此外,借助特定應用的評分函數為推理過程提供指導,Evo 2能夠設計出超越DNA本身的複雜生物架構。
Evo 2在預測「哪些突變是無害的」與「潛在的致病性」方面實現了超過90%的準確率,可以通過找到人類疾病的遺傳原因並加速新藥的開發,節省無數小時的研究時間和資金。
在未來,研究人員或許可以借助Evo 2的力量,解決人類那些「不治之症」,徹底消滅疾病。
Evo2架構:向光榮進化的矽基生物
Evo 2能夠對DNA序列進行建模,並在中心法則的各個層面實現應用,涵蓋分子和細胞尺度;模型基於DNA序列學習,無需針對特定任務進行微調,就能準確預測遺傳變異的功能影響,例如非編碼致病變異和具有臨床意義的BRCA1基因變異。
Evo 2採用新的多混合StripedHyena 2架構,展示了短顯式(SE)、中正則化(MR)和長隱式(LI)Hyena運算符的高效塊佈局,使Evo 2能夠使用比Evo 1使用多30倍的數據進行訓練,並且每次推理的核苷酸數量提升8倍。
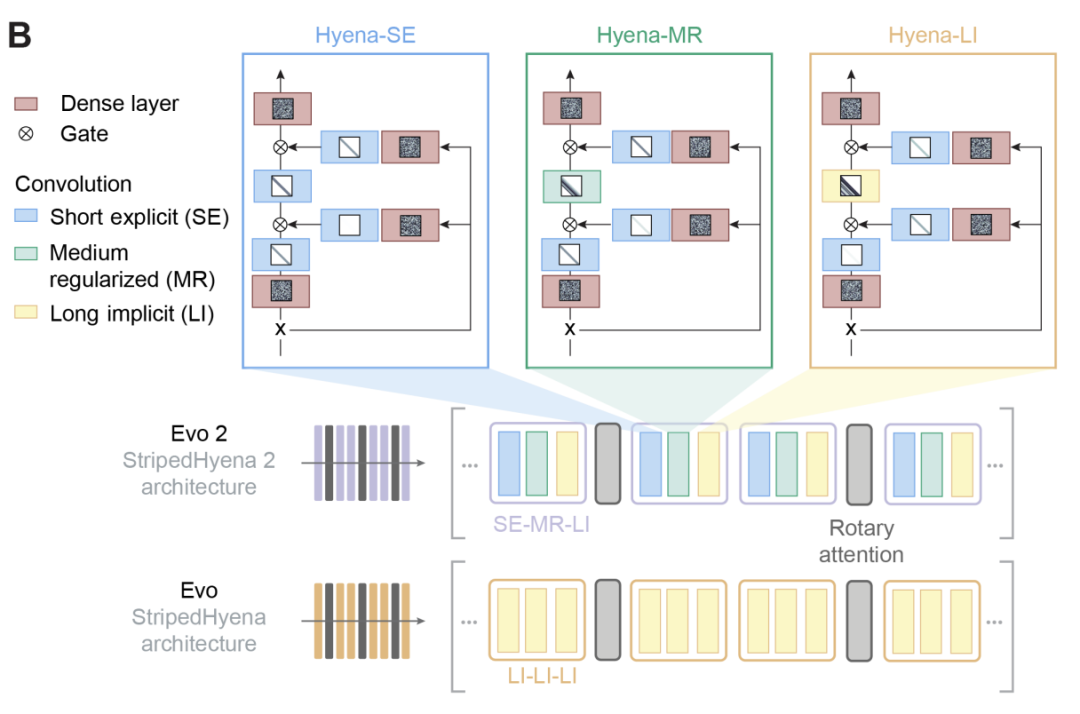
Evo 2採用「兩階段」訓練策略,在AWS上的NVIDIA DGX Cloud AI平台上經過幾個月的訓練,使用了超過2000塊NVIDIA H100 GPU,可以一次性處理高達100萬個核苷酸的長基因序列,使其能夠理解基因組中相隔較遠部分之間的關係。
在預訓練階段,Evo 2通過新穎的數據增強和權重分配方法,優先學習功能性的遺傳元素;在中訓練階段,則專注於長序列的構建。
Evo 2的40B和7B版本在訓練時分為短序列預訓練階段和長上下文中訓練階段。
在1024個GPU、40B規模下,StripedHyena 2相較於StripedHyena 1和Transformer架構,展現出更高的吞吐量。
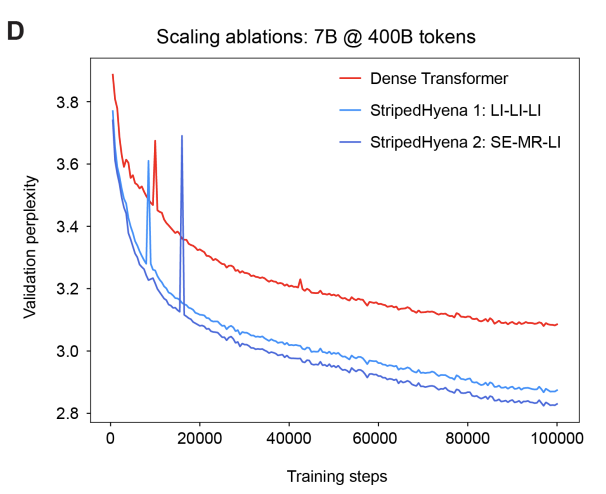
Evo 2的中訓練驗證困惑度表明,隨著模型規模和上下文長度的增加,模型性能得到提升。
通過修改後的「大海撈針」任務,Evo 2展示了其在長達100萬個序列長度的長上下文中進行有效回憶的能力。
實驗結果
通過在大規模的進化訓練數據集上學習序列的概率,生物序列模型能夠在沒有任何針對特定任務的微調或監督的情況下,瞭解突變效應與生物功能之間的相關性,即零樣本預測。
此前有效的零樣本突變效應預測僅在「僅用蛋白質序列」訓練的語言模型,或「僅用原核生物序列」訓練的基因組語言模型中得到證實。
Evo 2能夠在中心法則下的三種形式(DNA、RNA、蛋白質)和生命的三個領域(原核生物、古菌、真核生物)中學習序列可能性的分佈,因此研究人員評估了Evo 2是否能夠在所有這些形式和生物體中實現突變效應的預測。
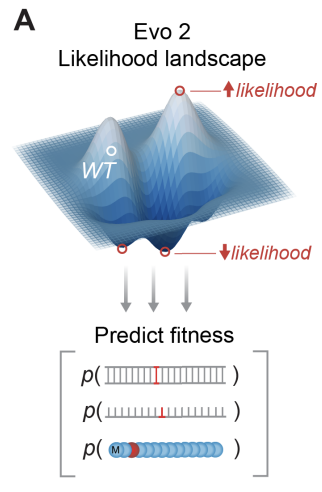
所有生命領域的編碼序列都遵循一個基本結構:以起始密碼子開始,以終止密碼子結束,並使用三聯密碼子來定義閱讀框架。
為了評估Evo 2是否掌握了這些基本的生物學原理,研究人員首先測試了單核苷酸變異(SNVs)對Evo 2在蛋白質編碼基因起始密碼子周圍基因組序列中的可能性的影響。他們在野生型序列的每個位置引入這些突變,並計算了Evo 2預測的可能性在數千個這樣的位點上的變化。
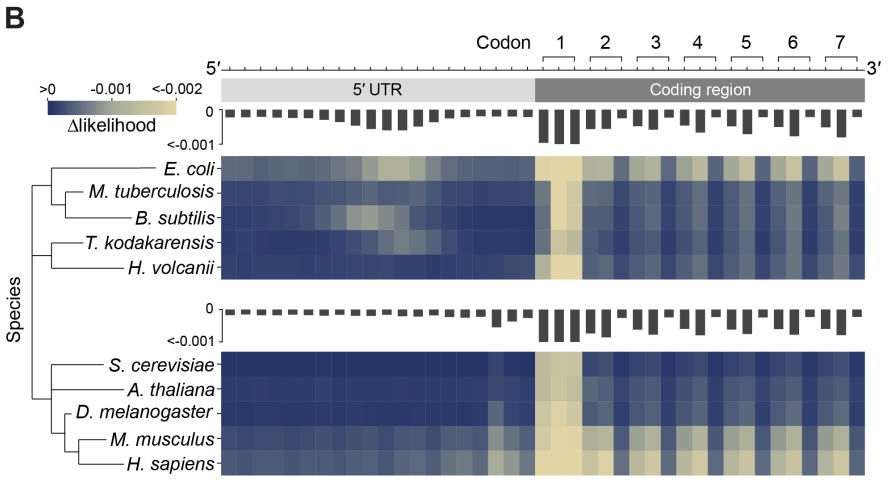
結果表明,在原核生物和真核生物中,起始密碼子內的突變會導致更劇烈的可能性變化,呈現出三聯密碼子的週期性模式,其中擺動位置的突變對可能性的影響較小。
在原核生物和真核生物的基因組中,研究人員還觀察到一個與保守的核糖體結合位點一致的模式,證實了模型已經學會了這些基本的遺傳特徵,儘管模型在訓練數據中從未見過這些序列的標註。
人類目前對基因組的理解認為,特定的遺傳變化應該導致不同的表型後果。
例如,錯義突變應該比同義突變更具破壞性,移碼突變和提前終止密碼子應該是最具破壞性的,而在必需的非編碼元件中,缺失的影響應該比在基因間區域的缺失更大。
通過測量非編碼和編碼序列中各種突變的影響,研究人員評估了Evo 2的概率是否能夠捕捉到這些已知的生物學規律。
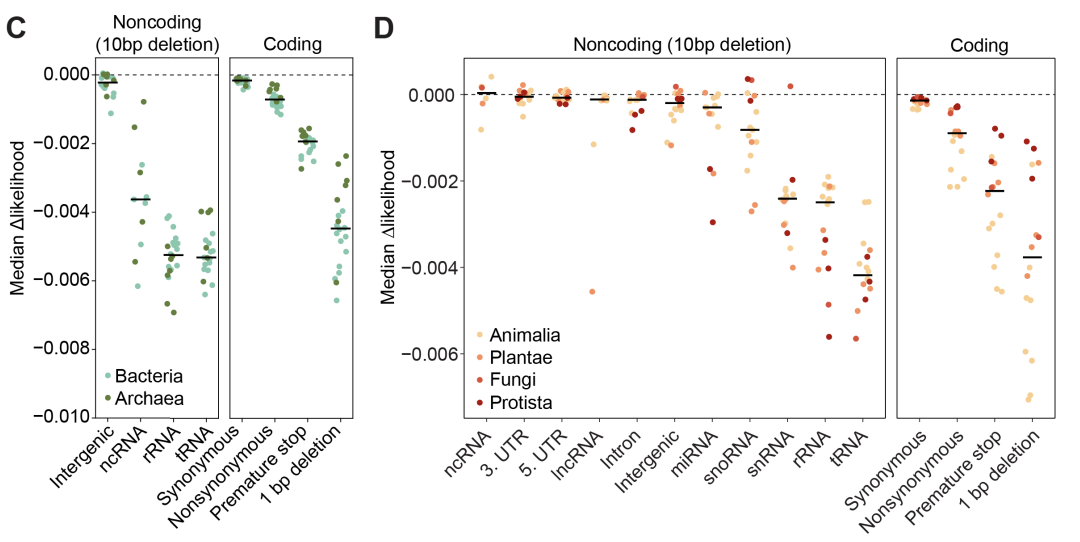
在20種原核生物和16種真核生物中,模型的概率變化與已知的生物學限制一致。
在編碼序列中,錯義變異、提前終止密碼子和移碼突變導致的可能性變化遠大於同義突變;
在非編碼區域中,tRNA和rRNA中的缺失比基因間區域和其他非編碼位點的缺失有更大的影響,符合對RNA重要性的已知信息。
未來,矽基智能創造細胞?
展望未來,研究人員設想了多種策略來提高Evo 2預測和設計的質量,目前可能更注重基因組序列的普遍進化分佈,而不是特定的分類學特徵。
將Evo 2與更多特徵和人類基因組變異數據相結合,可能有助於改善致病性預測或分析結構變異;
利用機制可解釋性,學習到的特徵還可以增強對更複雜生物學概念的檢測能力,並通過激活引導和特徵限制來指導模型生成,從而實現對生成結果的可編程控制;
為了提高Evo 2生成功能的質量,可能需要通過實驗反饋進行監督微調或強化學習;
Evo2初步證明了通過推理時計算設計複雜生物系統的概念,未來這種方法還可以擴展到包括其他特性,例如選擇性剪接、細胞類型特異性或基因回路功能。
進化論是生物學的統一理論,從基因到群體,通過DNA這一基礎信息層傳遞自然選擇的功能效應,Evo系列模型為生物學建模和設計奠定了基礎,將生物學中不同尺度的信息統一到一個共同的表徵中。
未來的工作如果將這一表徵與表觀基因組學和轉錄組學等更多模態信息相結合,可能會產生一個虛擬細胞模型,能夠有效地模擬健康和疾病中的複雜細胞表型。
參考資料:
https://x.com/pdhsu/status/1892243493445050606
https://x.com/MichaelPoli6/status/1892242976942035029